Description
1. Introduction
Les réseaux de neurones profonds constituent une approche prometteuse pour le développement d’outils d’aide à la décision dans le domaine de la santé. Cependant, l’utilisation de ces modèles nécessite une grande quantité de données d’entraînement pour atteindre des performances satisfaisantes. En outre, le processus d’acquisition des données est souvent problématique car il est soumis à la législation sur la protection des données.
Par conséquent, le domaine de l’imagerie médicale souffre d’un manque de données réelles (provenant de nos hôpitaux), ce qui entrave le développement d’outils d’aide aux soins de santé.
L’apprentissage fédéré est une solution pour surmonter cette disponibilité limitée de données. La brique D-Sail comprend des outils nécessaires pour entraîner un modèle global à partir de plusieurs sous-modèles. L’avantage principal est que les participants n’échangent pas leurs données locales tout en bénéficiant d’un modèle plus performant.
En échange, les partenaires doivent entraîner un modèle similaire sur leurs données locales. En particulier, dans l’apprentissage fédéré, le consortium d’acteurs partage les poids de leur modèle entraîné localement et une unité centrale les agrège.
2. Principe
La figure ci-dessous (Fig. 1) illustre le fonctionnement de l’apprentissage fédéré.
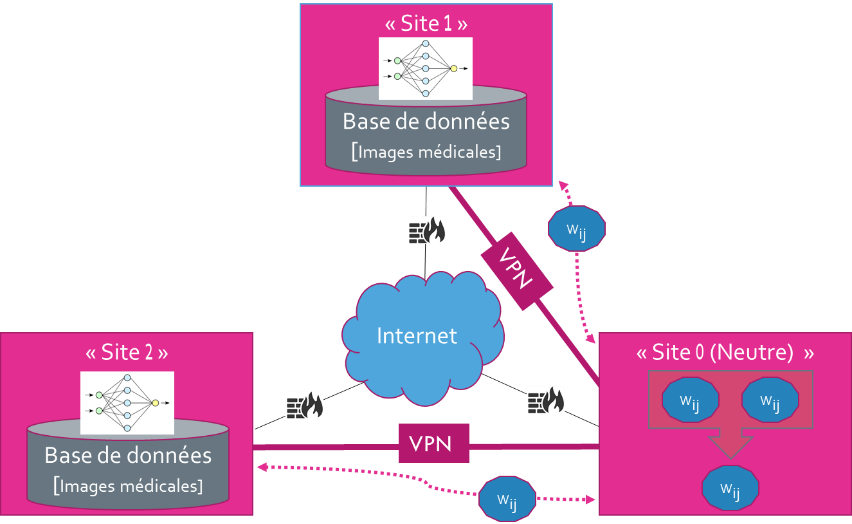
Fig. 1: Illustration du concept du Federated learning
Chaque site disposant de données (« Site 1 » et « Site 2 ») exécute son entraînement via des réseaux de neurones (directement sur ses propres données) sans que les données ne quittent l’institution (hôpitaux, centres de dépistage, industries, centres de recherche, universités, etc.) comme c’est généralement le cas avec l’entraînement traditionnel.
Seuls les poids résultant de l’entraînement des réseaux de neurones seront partagés avec les différents partenaires (« Site 1 » et « Site 2 »).
Une machine neutre (« Site 0 »), c’est-à-dire sans aucune donnée, joue le rôle unique d’agrégateur de poids. L’opération consiste donc à agréger les poids reçus et à les renvoyer à chaque partenaire afin de bénéficier de l’entraînement de chacun d’entre eux et ainsi permettre un entraînement de meilleure qualité grâce à son approche multicentrique et tout en respectant la confidentialité des données.
Pour éviter toute fuite éventuelle via les poids transmis à la machine neutre, le consortium s’appuiera sur un réseau privé (Virtual Private Network) afin d’assurer une communication sécurisée vers la machine neutre (« Site 0 »).
3. Méthode
Pour évaluer cette approche innovante, nous avons utilisé une base de données publique de 13 807 radiographies pulmonaires, composée de 10 192 images saines et de 3 615 images positives au COVID-19. Ces images anonymisées au format DICOM ont été converties au format PNG sans dégradation de l’image.
Ce jeu de données a été divisé en trois parties (non égales) afin de valider notre approche (apprentissage fédéré) avec la réalité du terrain, c’est-à-dire avec des jeux de données qui restent dans les hôpitaux.
Quant au modèle d’apprentissage automatique utilisé pour l’analyse des images de radiographie thoracique, il peut être reformulé comme une tâche de classification binaire d’images (positif/négatif). Cela constitue un problème de Machine Learning qui peut être traité efficacement avec la technologie basée sur le Deep Learning. Nous avons opté pour le réseau neuronal ResNet-18 pré-entraîné sur la base de données publique ImageNet.
4. Expérimentation
En pratique, les hôpitaux peuvent ne disposer que d’une quantité limitée de données, insuffisante pour entraîner efficacement un réseau de neurones compte tenu de la quantité limitée de données (notamment avec les cas positifs peu élevés et l’approche monocentrique). Ils peuvent également être réticents à partager des contenus sensibles avec d’autres hôpitaux. Nous avons émulé donc cette situation en considérant trois hôpitaux, chacun avec une partie mutuellement exclusive et équivalente du jeu de données original. L’apprentissage fédéré est utilisé, en agrégeant les poids issus des entraînements réalisés au sein de chaque hôpital, de manière itérative.
5. Interprétation des résultats
La courbe ROC (Receiver Operating Characteristic) illustrée ci-dessous (Fig. 2), également appelée courbe de sensibilité/spécificité, est utilisée pour calculer la capacité d’un test de dépistage à distinguer les personnes saines des personnes malades.
La sensibilité du test, c’est-à-dire les vrais positifs, est représentée sur l’axe des ordonnées et les faux positifs, c’est-à-dire la spécificité, sur l’axe des abscisses. La valeur optimale se trouve dans le coin supérieur gauche du graphique, ce qui indique une proportion élevée de vrais positifs et une faible proportion de faux positifs. L’aire sous la courbe (AUC) indique donc la précision du test : elle est égale à 1 si le test est parfait et peut identifier tous les patients sans faux positifs, à 0,5 si le test est sans valeur, détectant autant de vrais positifs que de faux positifs.
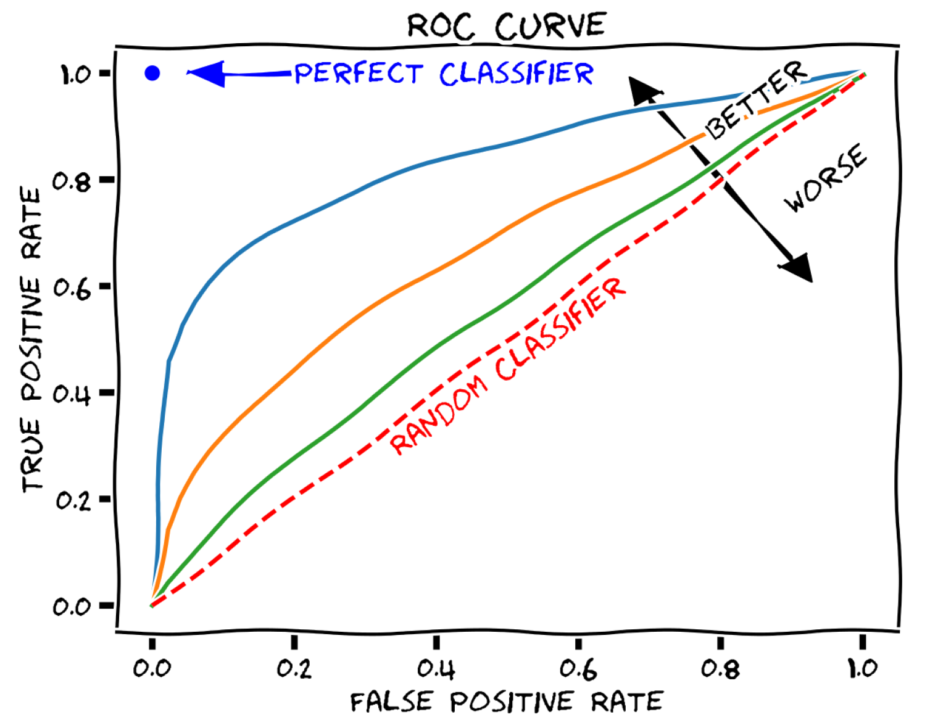
Fig. 2: ROC Curve
Comme le montre la figure ci-dessous (Fig. 3), les résultats sont très prometteurs, et nous avons obtenu d’excellents résultats avec ce jeu de données d’images thoraciques. La courbe rouge (H) obtenue avec notre solution est proche du point idéal situé dans le coin supérieur gauche.
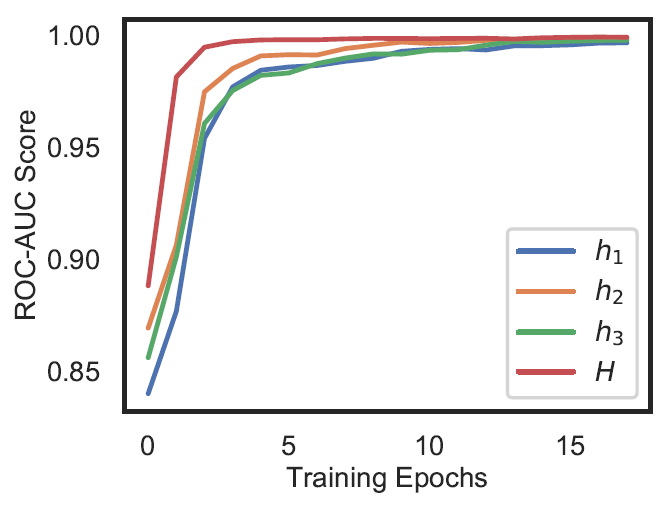
Fig. 3: Courbe ROC obtenue avec notre solution
6. Intégration de la brique D-SAIL dans la TRAIL Factory
La brique D-SAIL est basée sur une architecture conteneurisée gérée à l’aide d’un système Open Source de mise en oeuvre de conteneurs (Kubernetes) qui permet, à l’aide de scripts, de déployer une application, de la faire évoluer automatiquement.
Cette brique sera hébergée au CETIC au sein du cluster Kubernetes de la « TRAIL Factory ».
Un document richement illustré disponible ici explique également comment procéder à son installation sur Docker et Kubernetes sans passer par le cluster Kubernetes de la « TRAIL Factory ».
Lien vers le logiciel
https://github.com/XavierLessage/D-SAIL
Contributeurs
- Xavier Lessage, Senior Project Manager en Science des données au Centre de Recherche Agrée CETIC

